第 4 回のコラムでN-結合型糖鎖を紹介し、前回のコラムから「N-結合型糖鎖を解析する」という内容をスタートさせています。糖鎖のプロファイルを知るためには、試料からの糖鎖切り出し、精製やラベル化などの前処理、質量分析、データ解析を実施する必要があります。今回のコラムでは、タンパク質のN-結合型糖鎖プロファイル解析について簡単に紹介します。
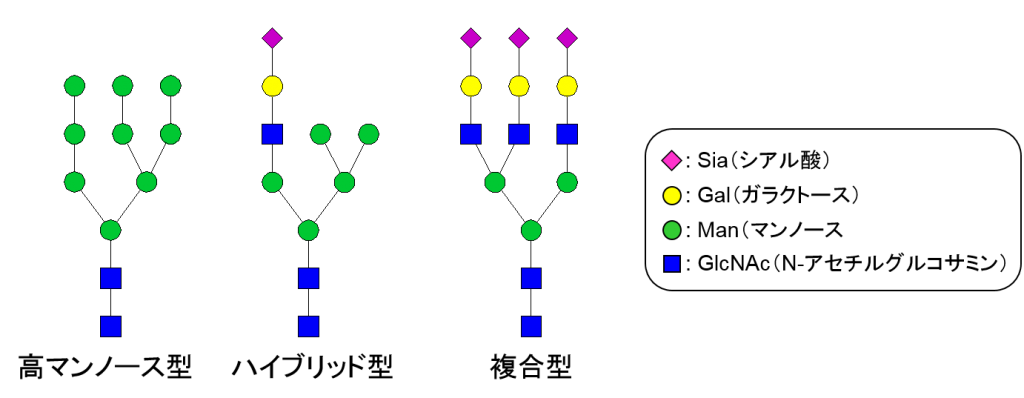
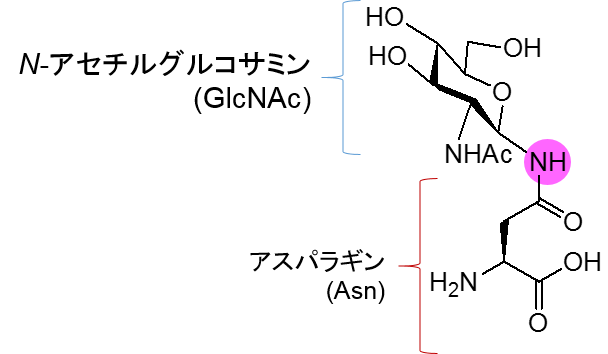
哺乳類におけるタンパク質のN-結合型糖鎖修飾は、下図のような糖鎖がアスパラギン側鎖に結合しています(抗体では 2分岐型のシンプルな糖鎖が多い)。
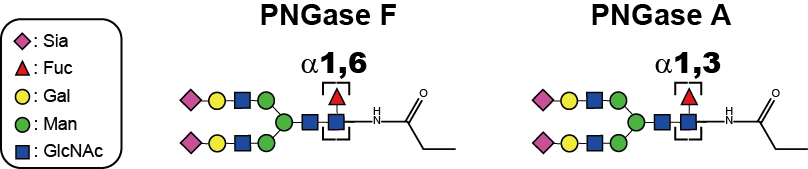
これらの糖鎖を解析するためには、タンパク質から糖鎖を切り出す必要があります。切り出す方法としては化学的手法、酵素法が考えられますが、N-結合型糖鎖の場合は便利な酵素が存在します。糖鎖脱離酵素である N-グリカナーゼ(PNGase)がその酵素です。糖鎖解析に使用されるPNGase には、PNGase F やPNGase A などがあり、目的に応じて使用されています。PNGase は温和な条件で、選択的にタンパクから N-結合型糖鎖を切り出すことが可能です(下図)。
PNGase F と A の使い分けですが、コアフコースの結合様式によって決まります。PNGase F は GlcNAc に結合した Fuc が α1,6結合の場合に、PNGase A はGlcNAc に結合した Fuc が α1,3結合の場合に選択されます。哺乳類由来のタンパク質のコアフコースは一般的にα1,6 結合が多く(疾患によってはα1,3も一部存在)、植物や昆虫などに由来するタンパク質にはα1,3結合が多いことからタンパクの由来によってどちらか一方、あるいは両方を使用します。
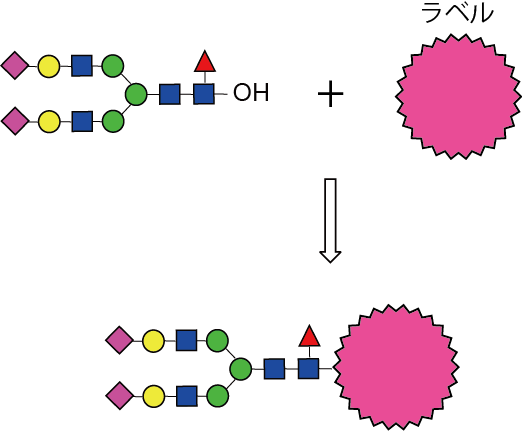
切り出された糖鎖はそのままではUV吸収が無いことと、検出感度を高めるために、アノマー位を任意の官能基でラベル化します。ラベル化には可逆的なものと、不可逆的なものがあり、質量分析の前にLCを実施するか否か(あるいは研究室、会社の伝統など)でラベル化剤を選定します。プロファイル解析の場合は、MALDI (Matrix Assisted Laser Desorption/Ionization) 型の装置を使用するためラベル化剤は任意のもので問題ありません。
PNGase の反応は選択性があることから、当該酵素処理によって切り出された糖鎖は N-結合型糖鎖のみとなります。よって、ラベル化された糖鎖を質量分析することで得られた一連の m/z 値に一致する組み合わせ(Hex、HexNAc、NeuAc、dHex )を計算することで、検体の糖鎖プロファイルを知ることができます。
以上が、大まかなプロファイル解析の流れとなります。今回、切り出された糖鎖の精製(捕捉)、シアル酸のカルボン酸修飾など質量分析前に必要な前処理には触れていませんが、実際良いデータを得るためにはこれらの前処理(糖鎖に限らず解析全般と同様に)が非常に重要な要素となってきます。
弊社では、熟練した技術と豊富な経験、知識を持った研究員による受託解析サービス、合成サービスを実施しております。熟練した技術に基づく分析サンプル調製、HPLC、質量分析、NMR などの機器を駆使することで、種々の目的に応じた解析に対応しています。受託合成においては、蓄積した各種知見、ノウハウなどを駆使し様々なターゲットの合成実績がございます。まずはお気軽に御相談ください。ご予算に応じてベストなご提案をさせていただきます。

筆者プロフィール
naruken
博士(理学)北海道大学大学院理学研究科
専門:糖鎖工学、タンパク質工学、構造解析